北京基因組所(國家生物信息中心)合作開發單細胞轉錄組數據仿真軟件SimCH
近十多年來,單細胞RNA測序技術(scRNA-seq)不斷發展和普及,同時數以千計的生物信息學/計算生物學工具被開發出來,以解決諸如數據獲取(包括量化、比對、拼接等)、數據清洗(包括質控、過濾、歸一化、整合等)、細胞分配(包括聚類、排序、分類等)、基因識別(包括基因網絡、差異表達、標記基因等)等scRNA-seq數據處理和分析步驟。盡管其中大部分軟件經過同行評議且在其論文中也彰顯了優點,但它們自己的比較可能存在偏好(bias),其可靠性尚未得到系統性檢測(benchmark)。
近年來出現了一些分別針對差異表達、聚類、插補、軌跡推斷、基因調控網絡推斷等分析的第三方系統性檢測,通常使用大量的實驗數據和仿真數據作為基準參考,來評估多個同類型方法在各項指標上的性能優劣。雖然實驗數據因其真實性較好,通常作為工具評估的“金標準”,但實驗數據的獲取和適合性也存在問題。相比較而言,仿真數據具有能快速產生、經濟、擴展性好等優點,可以作為工具評估的“銀標準”。近日,中科院北京基因組所(國家生物信息中心)張治華研究團隊與揚州大學孫磊副教授聯合在生物信息學著名期刊Briefings in Bioinformatics發表了題為SimCH: simulation of single cell RNA sequencing data by modeling cellular heterogeneity at gene expression level的研究型論文,推出了一款用于產生scRNA-seq仿真數據的軟件--SimCH。SimCH是一種半參數化(semi-parametric)的生成模型,它基于實驗數據估計統計學模型參數,并提供給用戶修改參數(如細胞數量、基因數量、測序深度、是否零膨脹)的接口,主要模式有SimCH-flex、SimCH-fit、SimCH-copula及擴展模式SimCH-ext。SimCH能通過高斯Copula模型在仿真數據中保留實驗數據的基因共表達信息。通過配置合適的參數,SimCH產生的仿真數據能夠很好地擬合同質性/異質性+UMI/非UMI技術的scRNA-seq實驗數據。實驗結果表明SimCH的整體性能優于現流行的仿真工具Splat和2021年推出的仿真工具scDesign2。
SimCH可應用于評估諸如細胞聚類、差異表達分析、軌跡推斷、插補等不同類型的計算方法性能。研究人員還發現,基因共表達信息與數據樣本的異質性密切相關。SimCH能夠有力支持scRNA-seq計算方法的系統性檢測,助力單細胞組學研究。
孫磊副教授為本文第一作者,孫磊和張治華研究員為共同通訊作者,碩士研究生王公銘參與了軟件開發。項目得到國家自然科學基金的資助。
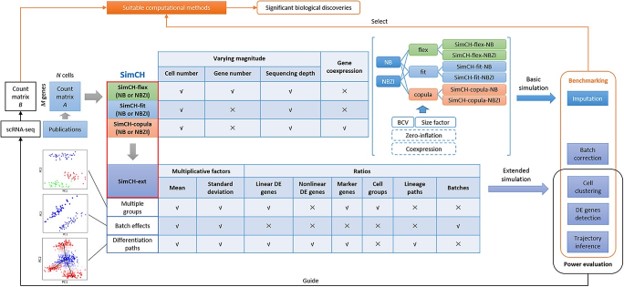
SIMCH運行框架圖