北京基因組所(國家生物信息中心)合作開發單細胞RNA m6A測序技術(scm6A-seq)
近日,中國科學院北京基因組研究所(國家生物信息中心)楊運桂研究員、中國農業大學韓建永教授、中國科學院北京基因組研究所(國家生物信息中心)楊瑩副研究員和鄭州大學第一附屬醫院孫瑩璞教授以共同通訊作者在Nature Communications 雜志上發表了題為“scm6A-seq reveals single-cell landscapes of the dynamic m6A during oocyte maturation and early embryonic development”的研究論文。該研究利用多重標記結合m6A-RIP-seq技術原理,開發了單細胞m6A測序技術(scm6A-seq), 并繪制了卵母細胞成熟和早期胚胎發育過程的單細胞分辨率m6A圖譜。
scm6A-seq技術實現了單細胞內轉錄組和m6A甲基化組的同時測序(圖1),在單個小鼠卵母細胞中可檢測到超過一萬個基因的表達,鑒定的m6A修飾區域呈現經典的RRAC保守基序特征,且技術重復性良好。通過與公共數據比較,該技術可在篩選到卵母細胞中保守的m6A修飾特征的同時,保留單細胞中的異質性修飾特征。
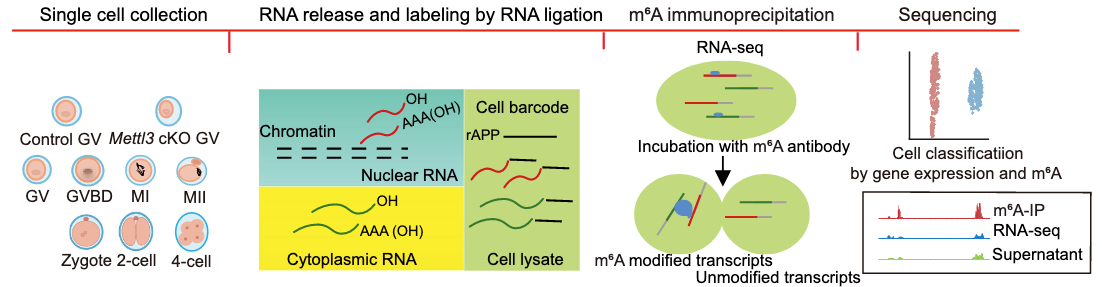
圖1. scm6A-seq測序方法和本研究得到的數據集
利用scm6A-seq對Mettl3敲除小鼠的卵母細胞RNA進行測序,發現Mettl3敲除的小鼠卵母細胞中m6A水平降低,同時METTL3依賴的m6A修飾基因在METTL3敲除的卵母細胞中出現異常積累。結合表達和m6A修飾特征進一步區分了高質量的卵子(SN)和低質量的卵子(NSN)(圖2)。
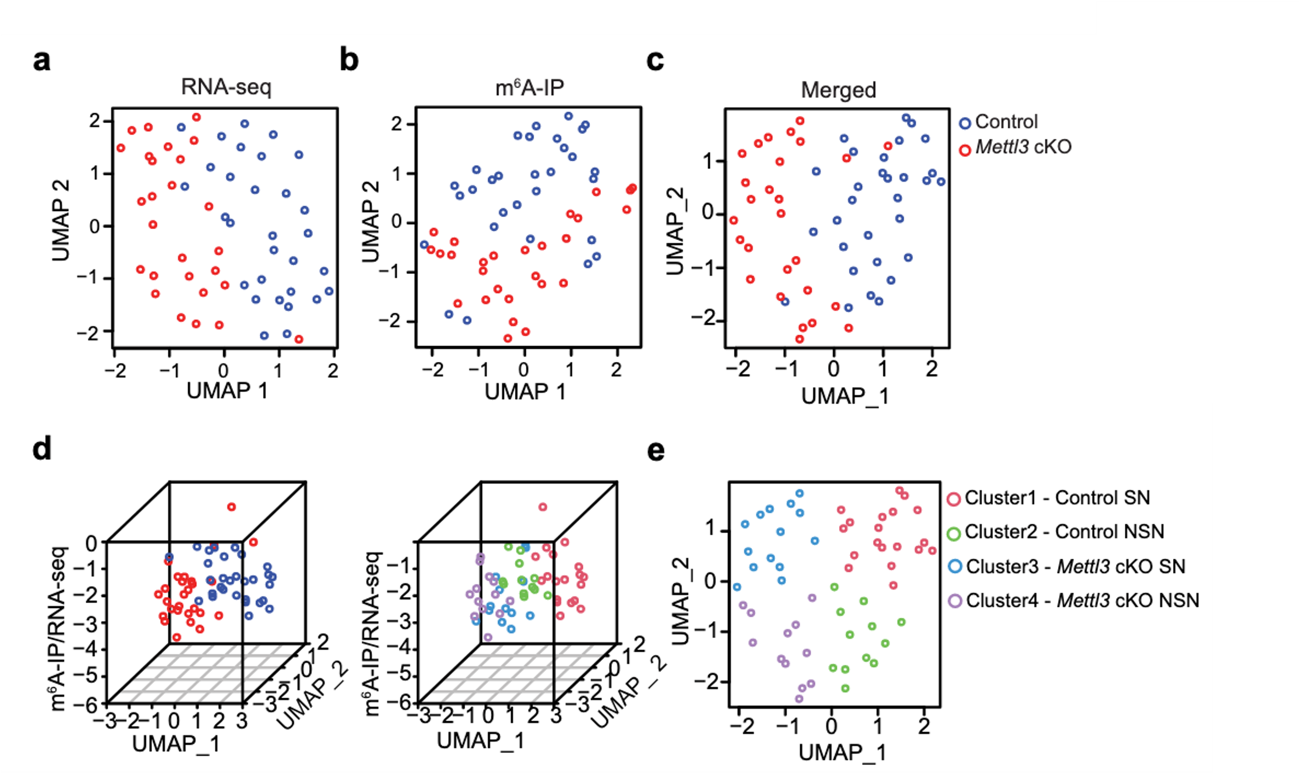
圖2. scm6A-seq區分SN和NSN卵母細胞
對不同減數分裂階段的卵子進行單細胞m6A測序,發現減數分裂恢復后m6A水平會出現明顯上升,呈現動態性修飾特征。結合對早期胚胎發育過程的單細胞m6A數據,發現m6A修飾的轉錄本在次級卵母細胞到合子轉變期間會更加穩定,而在Mettl3敲除的卵母細胞到合子的發育中,修飾轉錄本呈現不穩定性降解,提示在卵母細胞到合子發育期間m6A穩定母源修飾RNA的作用。在早期胚胎(合子到4細胞)發育過程中,發現2細胞胚胎的兩個卵裂球會出現基因組激活不同步現象,并鑒定到差異性m6A修飾的轉錄因子。
綜上,scm6A-seq技術實現了單細胞中m6A甲基化組和轉錄組的同時測序。利用scm6A-seq技術,鑒定出了高質量卵母細胞(SN)和低質量(NSN)卵母細胞,提示scm6A-seq技術具備更精確鑒定單細胞亞群的潛力。