北京基因組所(國家生物信息中心)發布動植物基因組變異與表型關聯知識庫GWAS Atlas v2.0
基因組序列變異與表型關聯知識,主要是通過全基因組關聯分析(GWAS),在全基因組范圍內鑒定與特定疾病或表型性狀等相關聯的遺傳變異位點,是挖掘和揭示生物復雜性狀分子遺傳機制的重要資源。
近日,中國科學院北京基因組研究所(國家生物信息中心)國家基因組科學數據中心正式發布更新版動植物基因組變異與表型關聯知識庫GWAS Atlas v2.0,以“GWAS Atlas: an updated knowledgebase integrating more curated associations in plants and animals”為題在國際學術期刊 Nucleic Acids Research 在線發表。
GWAS Atlas研究團隊每年持續審編公開發表的GWAS研究文章,按照統一的審編模型系統整理基因組變異與表型的關聯知識。GWAS Atlas v2.0基于830篇科研文獻,提供了15個物種(包括10種植物和5種動物)及與其1444個性狀相關聯的278109條基因型與表型關聯知識。為進一步精細定位與復雜生物性狀相關聯的候選遺傳位點,研究團隊通過對所有關聯知識進行連鎖不平衡分析,鑒定到與439個性狀相關的6084個獨立變異位點(Lead SNPs);并通過生物審編,新收錄157個性狀的486個經實驗驗證的因果變異(Casual variants)。這些遺傳位點的系統分析和整合可以幫助我們更好地理解復雜性狀的遺傳結構,為進一步高效定向育種提供更加精確的數據知識,進而提高種質資源利用率。
為更好服務用戶數據的匯交和分析,GWAS Atlas v2.0還建立了GWAS關聯知識的在線匯交功能,以方便用戶在線批量遞交研究結果,并為每一個遞交數據分配唯一可識別的標識符,根據遞交用戶設定的數據公開時間進行可控管理;同時新研發上線了LeadSNPFinder、GeneFinder、MHPlotter和QQPlotter四個在線分析和畫圖工具,方便用戶開展數據的挖掘應用分析和可視化分析結果。總之,GWAS Atlas 2.0為未來重要農藝性狀的模塊化遺傳研究和育種應用提供了重要數據管理和分析平臺。
北京基因組所(國家生物信息中心)博士研究生劉曉楠、工程師田東梅和李翠萍為本文共同第一作者,宋述慧研究員與章張研究員為共同通訊作者。該研究得到了中科院戰略性先導科技專項、國家重點研發計劃、國家自然科學基金、中科院青促會等項目資助。
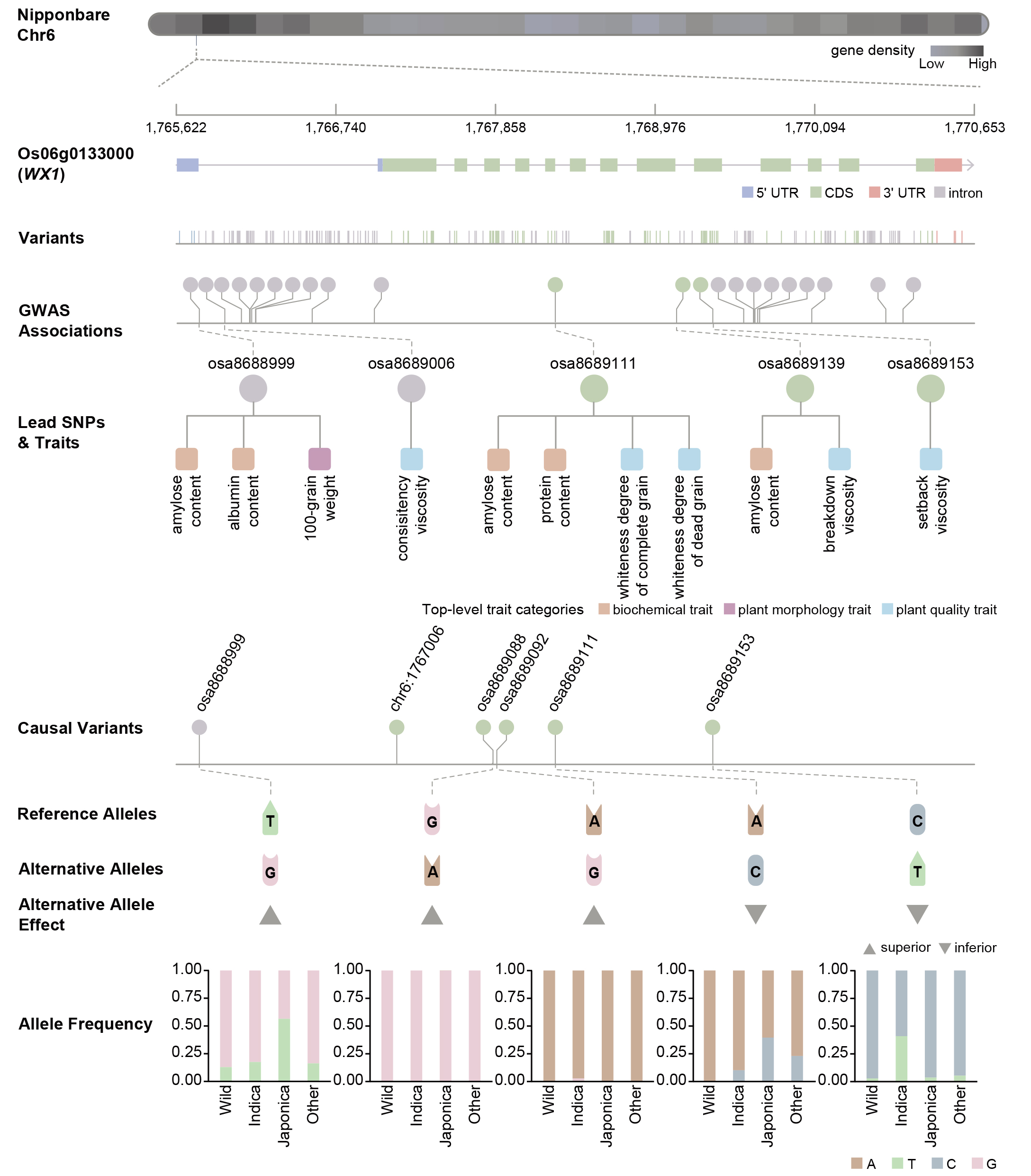
GWAS Atlas 數據示例,水稻WX1基因






