北京基因組所(國家生物信息中心)合作發現冠心病非脂質通路分子亞型致病新基因
冠心病是嚴重危害人類健康的常見心血管疾病,也是導致全球人類死亡的主要原因之一。這一疾病受基因組常見變異和罕見變異的共同復雜影響,遺傳因素對疾病的作用程度約為50%~70%。解析冠心病發生背后的遺傳基礎和機制,是依據基因組醫學對疾病進行精準分型,進而依據遺傳致病因素對疾病進行個性化治療的基礎。隨著測序技術的快速發展和成本的降低,大規模人群測序數據的產生使得深入解析冠心病遺傳基礎成為可能。
為進一步識別冠心病相關致病基因以及開展基于基因遺傳信息的疾病風險預測,北京基因組研究所(國家生物信息中心)汪敏先研究組與博德研究所(Broad Institute of MIT and Harvard)Amit V. Khera研究組展開合作研究,基于4.1萬例冠心病患者和21.7萬例對照組的全外顯子組基因測序數據和心血管疾病相關臨床風險因素等疾病電子病歷數據,開展基于基因編碼區功能變異的關聯富集分析(圖1),發現內皮一氧化氮合成酶基因(NOS3)為冠心病的非脂質分子亞型。相關研究成果以“Gene Sequencing Identifies Perturbation in Nitric Oxide Signaling as a Nonlipid Molecular Subtype of Coronary Artery Disease”為題在國際期刊Circulation: Genomic and Precision Medicine發表。
該研究基于超大規模的樣本量更加有效地檢測出基因編碼區上的罕見變異,這些編碼蛋白質的罕見變異能夠改變蛋白質表達、結構或功能,對疾病風險產生較大影響。同時,該研究將位于同一個基因上的所有罕見變異根據功能影響類型聚為一個集合,比較該基因上的變異總頻率在患病組和對照組之間的差異(圖1),該方法能夠高效分析基因與疾病表型之間的因果關系,克服了傳統關聯分析研究(GWAS)雖能定位基因組區域,但無法精確定位致病基因的缺點,從而為下游實驗機制驗證及藥物開發提供了遺傳機制明晰的靶點。
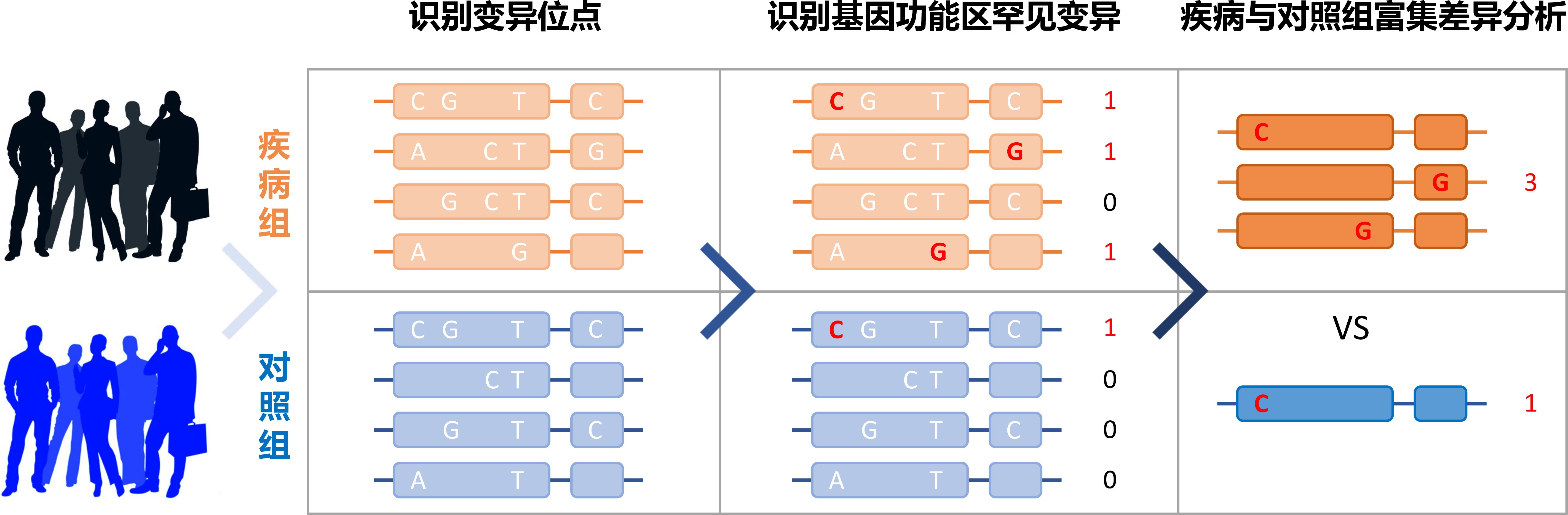
圖1. 通過分析基因編碼區功能變異富集差異發現致病風險基因的流程示意圖
該研究鑒定出低密度脂蛋白受體基因(LDLR,與低密度脂蛋白膽固醇代謝相關)的罕見變異與冠心病最密切相關(圖2)。在0.34%的對照組中發現LDLR變異,然而在0.91%的冠心病患者中發現了LDLR變異,矯正年齡、性別等混雜因素的影響后,攜帶者的患病風險增加了4.4倍。同時,該研究還發現了一種與LDLR不同的非脂質分子亞型,即與內皮一氧化氮合成酶基因(NOS3)相關的遺傳變異,是冠心病風險的另一個非常重要的驅動因素(圖2),0.59%的冠心病患者存在NOS3變異,對照組為0.41%,矯正混雜因素后,基因變異攜帶者的患病風險增加了約2.4倍。利用中間分子表型對疾病致病的機制解析表明,NOS3主要通過影響血壓增加冠心病風險,與傳統的血脂相關通路無顯著相關性。
該研究由中科院北京基因組研究所(國家生物信息中心)、博德研究所(Broad Institute of MIT and Harvard)、哈佛大學醫學院、美國麻省總院等機構合作完成。博德研究所Amit V. Khera教授與北京基因組研究所(國家生物信息中心)汪敏先研究員為并列第一作者,博德研究所Amit V. Khera教授為本研究的通訊作者。
為進一步增加該研究方法的統計效力,合作研究團隊正在對近5萬冠心病患者及近51萬的對照數據進行新一輪升級分析,從增大樣本量和構建分析新方法、新工具等多個維度,提升發現致病新基因的能力。同時,研究組希望和更多單位開展合作,共同推進對冠心病等心腦血管疾病遺傳基礎和機制的研究,促進對心腦血管疾病的精準干預和個性化治療。
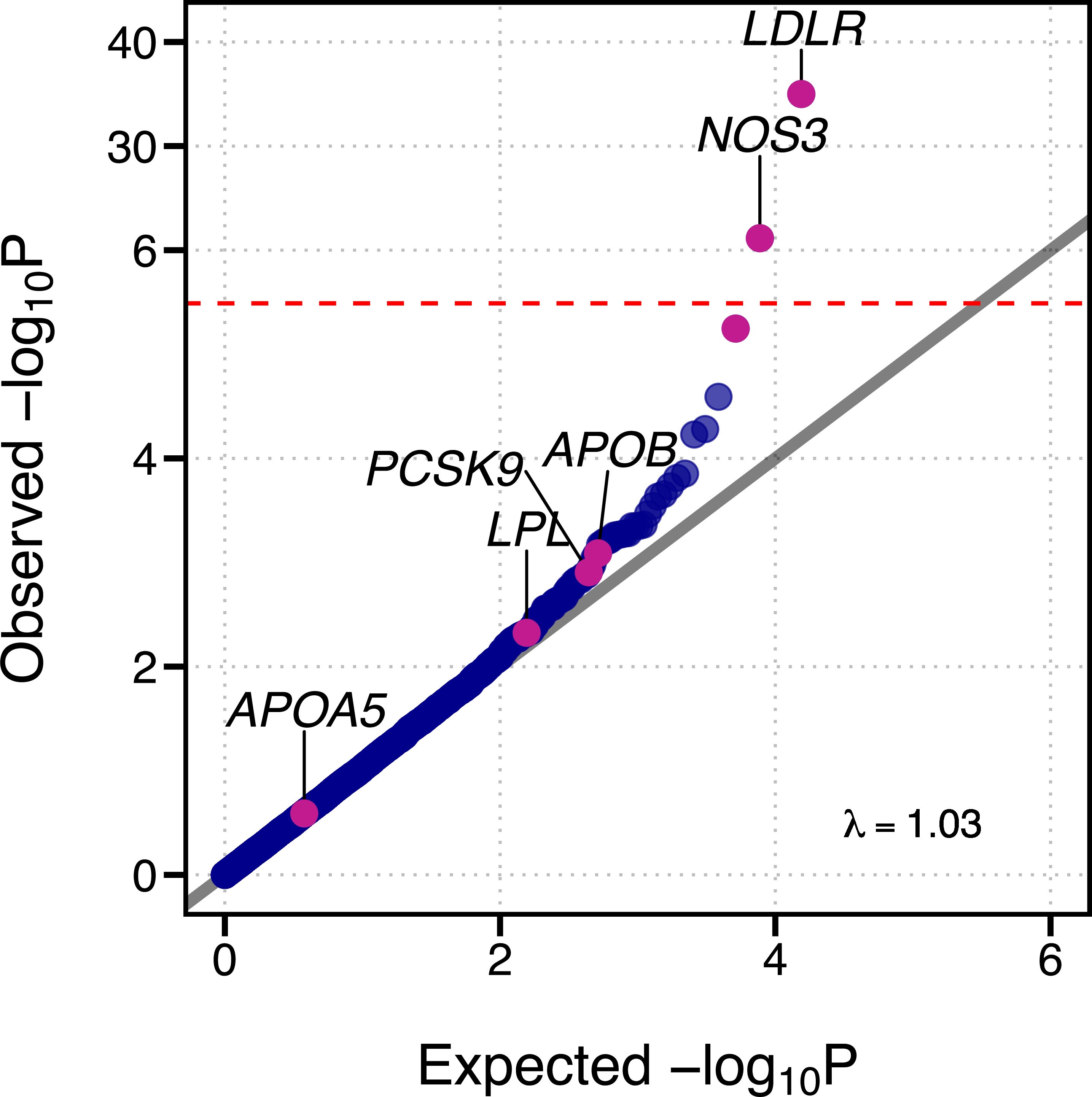
圖2. 依據基因突變在疾病與對照組間的富集差異程度排序的基因列表。紅色虛線表示全基因組顯著水平,P Value = 2.5x10-6